SEKWENCJONOWANIE PRZESTRZENNE TRUE SINGLE-CELL
30 Lipca 2025
SEKWENCJONOWANIE PRZESTRZENNE
TRUE
SINGLE-CELL
- Sekwencjonowanie przestrzenne, czyli kolejna przełomowa rewolucja w biologii molekularnej
Współczesna biologia molekularna nieustannie poszukuje nowych narzędzi, które pozwalają jeszcze dokładniej zrozumieć, jak działają komórki i tkanki. Jednym z najbardziej przełomowych osiągnięć ostatnich lat jest sekwencjonowanie przestrzenne (ang. spatial sequencing), czyli technika, która pozwala nie tylko odczytać, jakie geny są aktywne w danym fragmencie tkanki – ale też gdzie dokładnie w przestrzeni ta aktywność zachodzi. To nic innego jak połączenie analizy molekularnej z „mapą tkanek” o rozdzielczości komórkowej lub subkomórkowej.
Na długo przed pojawieniem się metod NGS, naukowcy próbowali zrozumieć, gdzie w komórce lub tkance znajduje się dany RNA lub białko. Narzędzia, które stosowano to mi.in:
- Immunohistochemia (IHC) – wykrywanie białek za pomocą przeciwciał.
- In situ hybridization (ISH) – hybrydyzacja sond do RNA bezpośrednio na skrawkach tkanek.
- FISH (Fluorescence in situ hybridization) – wersja fluorescencyjna ISH, szczególnie używana w cytogenetyce.
Niestety, ww. metody miały jedno zasadnicze ograniczenie, pozwalały badać tylko kilka genów jednocześnie, a analiza była czasochłonna i niskoprzepustowa. Wraz z rozwojem i popularyzacją metod sekwencjonowania metod nowej generacji (NGS) powszechne stało się masowe sekwencjonowanie RNA (RNA-seq) oraz analiza całego transkryptomu. Jednak klasyczne RNA-seq usuwało informację przestrzenną, ponieważ RNA było ekstrahowane z całej tkanki jako całość. W odpowiedzi na tę lukę, rozpoczęto badania nad metodami, które pozwalałyby „dodać kontekst przestrzenny” do analizy RNA. W latach 2006-2010 zespół pod kierownictwem Prof. Joakima Lundeberga i Sten Linnarssona w Szwecji, na Karolinska Institutet, opracował pierwsze metody dołączenia „znacznika przestrzennego” do RNA w tkance. Jednakże przełomem w tej dziedzinie okazała się być publikacja Ståhl i wsp., "Visualization and analysis of gene expression in tissue sections by spatial transcriptomics" [1], która ukazała się w 2016 w Science. Opisuje on pierwszy zautomatyzowany system spatial transcriptomics pozwalający na analizę RNA w skrawkach tkanek za pomocą „adresowanych” oligonukleotydów na szkiełku. W ten sposób możliwe stało się sekwencjonowanie RNA z zachowaniem informacji o jego położeniu w tkance.
W 2018 firma 10x Genomics przejęła Spatial Transcriptomics AB i opracowała platformę Visium Spatial Gene Expression. Pomimo wysokiej przepustowości i późniejszej ewolucji platformy do visium HD (high definitione) platforma ta ma pewne ograniczenia wynikające z relatywnie niskiej rozdzielczości ∼55 µm (jedna plamka = kilka komórek). Nowa generacja technologii, która umożliwia bezpośrednie wizualizowanie i identyfikację transkryptów mRNA in situ, w niezmienionych tkankach, bez potrzeby ekstrakcji RNA i bez sekwencjonowania NGS. Przykładem jest rozwiązanie 10x Genomics – Xenium. Xenium to system oparty na cyfrowym obrazowaniu fluorescencyjnym z sondami hybrydyzującymi do mRNA in situ, oferujący m.in. rozdzielczość subkomórkową. W tym przypadku ograniczeniem jest brak możliwości analizy całego transkryptomu.
|
Visium (kodowanie + NGS) |
Xenium (obrazowanie RNA) |
|
|
Technika |
Hybrydyzacja + sekwencjonowanie NGS |
Cykliczna hybrydyzacja fluorescencyjna |
|
Rozdzielczość |
∼55 µm (jedna plamka = kilka komórek) |
Subkomórkowa (∼0.5 µm) |
|
Liczba genów |
∼20,000 (bulk RNA-seq) |
do 1000–2000 (wybór targetów) |
|
Odczyt |
NGS (Illumina) |
Bezpośredni obraz |
|
Próbki |
Świeżo zamrożone, FFPE |
FFPE, mrożone |
- Trekker – czyli prawdziwy single-cell spatial sequencing
Takara Bio w swojej ofercie posiada rewolucyjne rozwiązanie Trekker, które dzięki swojej rozdzielczości (∼10 µm) pozwala na pierwsze, prawdziwe sekwencjonowanie przestrzenne pojedynczych komórek - real single-cell spatial sequencing.
Trekker działa poprzez znakowanie każdego jądra w jego naturalnym środowisku tkankowym unikalnymi, przestrzennymi kodami kreskowymi. Te przestrzenne barcody są odczytywane za pomocą sekwencjonowania nowej generacji (NGS), co pozwala na bioinformatyczne umiejscowienie każdego jądra w jego współrzędnych przestrzennych. Rezultatem jest mapa przestrzenna z rzeczywistą rozdzielczością pojedynczej komórki bez użycia skomplikowanej aparatury, dekonwolucji typu komórki czy segmentacji komórek. Twórcy technologii Russell i wsp. [2], opublikowali wyniki otrzymane z wykorzystaniem tej przełomowej technologii w Nature w 2024 roku. Początkowo nazywając Trekker – slide tags.
|
Curio Trekker |
10x Genomics Visium |
Xenium / CosMx |
|
|
Rozdzielczość |
~10 µm (single-molecule) |
~55 µm (wielokomórkowa) |
~0,5 µm (subkomórkowa) |
|
Potrzeba specjalistycznego sprzętu |
nie |
tak |
tak |
|
Szybkość przygotowania |
1 dzień |
2–3 dni |
kilka dni |
|
Analiza dużych tkanek |
tak (cały przekrój) |
tak |
ograniczona do ROI |
|
Wymagane działania w analizie danych |
brak |
dekonwolucja |
segmentacja |
Poza unikatową rozdzielczością, rozwiązanie Trekker charakteryzuje się brakiem konieczności inwestowania w kosztowe urządzenia. Protokół jest w pełni kompatybilny z większością sekwenatorów single-cell. Konieczna jest jedynie integracja wyników. Takara Bio posiada zwalidowane protokoły pozwalające na integrację Trekker m.in. z systemem 10x Genomix Chromium. Rysunek 1 przedstawia uproszczony schemat pracy z systemem Trekker.
Jeśli chcesz dowiedzieć się więcej o systemie Trekker to odwiedź stronę stronę Takara Bio lub skontaktuj się z nami!
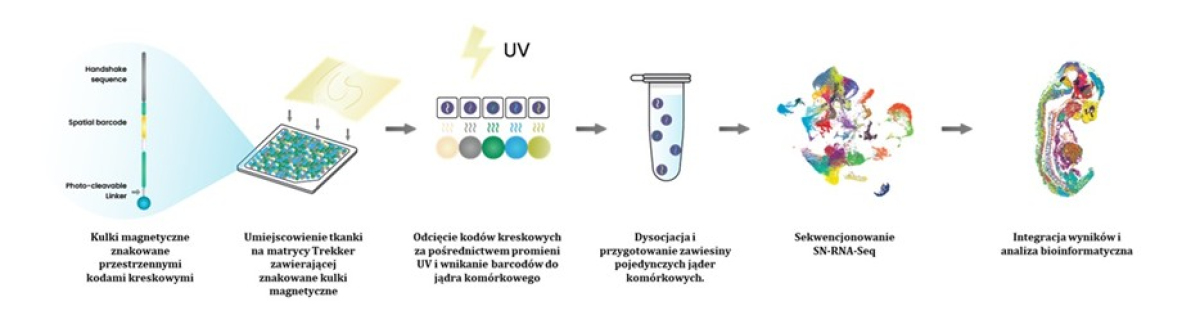
Rysunek 1. Uproszczony schemat pracy z systemem Trekker.
[1] Ståhl PL, Salmén F, Vickovic S, Lundmark A, Navarro JF, Magnusson J, Giacomello S, Asp M, Westholm JO, Huss M, Mollbrink A, Linnarsson S, Codeluppi S, Borg Å, Pontén F, Costea PI, Sahlén P, Mulder J, Bergmann O, Lundeberg J, Frisén J. Visualization and analysis of gene expression in tissue sections by spatial transcriptomics. Science. 2016 Jul 1;353(6294):78-82. doi: 10.1126/science.aaf2403. PMID: 27365449
[2] Russell AJC, Weir JA, Nadaf NM, Shabet M, Kumar V, Kambhampati S, Raichur R, Marrero GJ, Liu S, Balderrama KS, Vanderburg CR, Shanmugam V, Tian L, Iorgulescu JB, Yoon CH, Wu CJ, Macosko EZ, Chen F. Slide-tags enables single-nucleus barcoding for multimodal spatial genomics. Nature. 2024 Jan;625(7993):101-109. doi: 10.1038/s41586-023-06837-4. Epub 2023 Dec 13. Erratum in: Nature. 2024 Jan;625(7994):E11. doi: 10.1038/s41586-023-06961-1. PMID: 38093010; PMCID: PMC10764288.