Zrozumieć klonowanie molekularne: Podstawowe narzędzie w inżynierii genetycznej
21 Lutego 2024 (Ostatnia aktualizacja 17.10.2024)
Z wpisu dowiesz się:
- Co to jest klonowanie?
- Na czym polegało klonowanie owcy Dolly?
- Na czym polega klonowanie tzw. klasyczne z użyciem restryktaz i ligaz?
- Nomenklatura -podstawowe pojęcia (plazmid, insert, α-komplementacja)
- Czym jest klonowanie tzw. „seamless”? Przykłady systemów seamless
- Czym jest klonowanie In-Fusion®?
Klonowanie molekularne (molecular cloning) jest podstawową techniką inżynierii genetycznej, ułatwiającą manipulację i analizę cząsteczek DNA, prowadzenie replikacji elementów genetycznych oraz wprowadzanie egzogennej ekspresji. Proces ten, zapoczątkowany w latach 70. ubiegłego wieku, zrewolucjonizował różne dziedziny, od badań podstawowych po biotechnologię i medycynę. W swojej istocie klonowanie molekularne polega na replikacji, powieleniu fragmentów DNA w celu wygenerowania wielu jego identycznych kopii. Następnie w wyniku podziałów zmodyfikowanego organizmu jednokomórkowego dochodzi do powstania wielu identycznych klonów (np. szczepów bakteryjnych). Takie manipulacje umożliwiają naukowcom precyzyjne badania i analizy genetyczne.
Proces rozpoczyna się od przygotowania i izolacji interesującego fragmentu DNA. Często odbywa się to poprzez PCR (odczynniki PCR i qPCR) sekwencji z organizmu dawcy, takiego jak bakterie, rośliny lub zwierzęta. Fragment ten zawiera gen lub sekwencję, którą naukowcy chcą manipulować lub badać. Techniki izolacji DNA różnią się w zależności od źródła i złożoności materiału genetycznego, ale zazwyczaj obejmują trawienie enzymatyczne i etapy oczyszczania.
Po wyizolowaniu, fragment DNA jest wstawiany do wektora, cząsteczki DNA zdolnej do samoreplikacji w organizmie gospodarza. Wektory służą jako nośniki do przenoszenia obcego DNA do organizmu gospodarza, umożliwiając jego propagację i ekspresję. Powszechnie stosowane wektory obejmują plazmidy, które są kolistymi cząsteczkami DNA występującymi w bakteriach.
Kolejnym kluczowym etapem klonowania molekularnego jest ligacja fragmentu DNA do wektora. W procesie tym zazwyczaj pośredniczy ligaza DNA, enzym katalizujący tworzenie wiązań fosfodiestrowych między fragmentami DNA, tworząc rekombinowaną cząsteczkę DNA. Powstały konstrukt, znany jako rekombinowany wektor, zawiera obcy fragment DNA zintegrowany z jego genomem.
Po utworzeniu rekombinowanego wektora jest on wprowadzany do organizmu gospodarza w procesie zwanym transformacją. W przypadku bakterii, na przykład, często wiąże się to z wystawieniem komórek bakteryjnych na działanie roztworu zawierającego rekombinowany wektor, a następnie inkubacją w określonych warunkach, aby umożliwić pobranie obcego DNA.
Po transformacji naukowcy stosują różne techniki selekcji w celu identyfikacji komórek zawierających pożądane rekombinowane DNA. Mogą one obejmować markery oporności na antybiotyki lub inne geny reporterowe zintegrowane z wektorem, co pozwala na identyfikację i izolację transformowanych komórek. Po wyizolowaniu, komórki te mogą być hodowane w celu wygenerowania populacji klonów zawierających rekombinowane DNA.
Możemy zatem wyróżnić następujące etapy klonowania:
⦁ Otrzymanie fragmentu DNA zawierającego pożądany gen
⦁ Łączenie pozyskanych fragmentów z cząsteczkami wektorowymi
⦁ Wprowadzenie zrekombinowanych wektorów do komórek, w których mogą się powielać
⦁ Identyfikację i selekcję organizmów z pożądaną sekwencją genetyczną
W badaniach podstawowych klonowanie molekularne umożliwia badanie funkcji, regulacji i ekspresji genów. W biotechnologii klonowanie molekularne służy jako istotne narzędzie do produkcji rekombinowanych białek, szczepionek i innych biofarmaceutyków. W medycynie klonowanie molekularne ułatwiło rozwój technik terapii genowej w leczeniu zaburzeń genetycznych i nowotworów.
Na czym polegało klonowanie owieczki Dolly?
Zazwyczaj słowo klonowanie, kojarzy nam się ze słynną owcą Dolly. Procedura ta jednak różniła się znacznie od tej, którą zwykle określamy tym mianem i jaką się przeprowadza w laboratoriach biologicznych na całym świecie.
Wspomniany eksperyment klonowania był o tyle wyjątkowy, że po raz pierwszy pokazano, że ssak może zostać „odtworzony” (sklonowany) z komórki somatycznej organizmu. Zespół naukowców z Roslin Institute, pod przewodnictwem Iana Wilmuta i Keitha Campbella dokonało transferu jądra komórkowego diploidalnej (2n) komórki gruczołu mlekowego dorosłej owcy (SCNT- Somatic Cell Nucleus Transfer) do komórki jajowej (haploidalnej, 1n) pozbawionej jądra komórkowego (0n). Po tej manipulacji zaindukowano komórkę do podziałów w wyniku czego powstał zarodek, który następnie został wszczepiony do macicy owcy zastępczej. Tam rozwijał się aż do narodzin owieczki Dolly. Sklonowanie owcy Dolly w 1996 roku stanowiło niezaprzeczalnie kamień milowy w biotechnologii, oraz wpłynęło na dalszy rozwój genetyki i medycyny regeneracyjnej. W klasycznym rozumieniu otrzymaliśmy
Wróćmy jednak do metod bardziej klasycznego klonowania.

Fig1. Zdjęcie Iana Wilmuta z owieczka Dolly [źródło gazeta wyborcza].
Na czym polega klonowanie tzw. klasyczne z użyciem restryktaz i ligaz?
Klonowanie za pomocą enzymów restrykcyjnych jest podstawową techniką stosowaną w biologii molekularnej. Procedura opiera się na wykorzystaniu enzymów restrykcyjnych (restriction enzymes), znanych również jako restryktazy lub endonukleazy restrykcyjne, które są białkami rozpoznającymi i tnącymi obie nici DNA w określonych krótkich sekwencjach (ok 6-8 nt) znanych jako miejsca restrykcyjne. Przecinając DNA w tych precyzyjnych miejscach, enzymy restrykcyjne generują fragmenty z tzw. „lepkimi końcami” (sticky ends) lub „tępymi końcami” (blunt ends), w zależności od użytego enzymu. Końce te mogą następnie łączyć się w pary zasad z komplementarnymi sekwencjami na innych fragmentach DNA, czyli w plazmidzie.
Kolejnym krokiem w klonowaniu enzymami restrykcyjnymi jest ligacja (ligation) lub łączenie tych fragmentów DNA. Zazwyczaj odbywa się to przy użyciu ligazy DNA (ligase), enzymu, który ułatwia tworzenie wiązań fosfodiestrowych między sąsiednimi nukleotydami fragmentów DNA, skutecznie „sklejając” czyli ligując kawałki razem. Nowo utworzoną cząsteczkę rekombinowanego DNA można następnie wprowadzić do organizmu gospodarza, w procesie zwanym transformacją. Zwykle wykorzystuje się komórki bakterii szczepów E.coli tzw, kompetentnych (compitent cells) czyli tak modyfikowanych aby z łatwością pobierały elementy genetyczne. W organizmie gospodarza rekombinowane DNA może być replikowane lub eksprymowane w celu zbadania funkcji genu lub wytworzenia interesujących białek.
Ta metoda klonowania jest bardzo wszechstronna i odegrała kluczową rolę w dziedzinie genetyki, biotechnologii i medycyny, umożliwiając rozwój organizmów modyfikowanych genetycznie, terapii genowej i produkcji farmaceutyków. Niesie jednak ze sobą szereg ograniczeń, które można pokonać sięgając po tzw. metody „seamless” czyli bezszwowe o czym w dalszej części.
Podczas klonowania z użyciem enzymów restrykcyjnych jesteśmy zależni od działania enzymów – restryktaz i ligazy.
Przygotowanie insertu odbywa się na takiej zadzie, że przed i za naszą sekwencją musimy umieścić miejsca cięcia dla restryktaz. Zazwyczaj wykonuje się to za pomocą PCR. Miejsca te ponad to muszą być zbieżne do tych występujących w MCS plazmidu. Jednocześnie sama nasza wstawka nie może w swojej strukturze zawierać takiego miejsca, aby podczas trawienia końców insertu sama nie została zdeintegrowana.
Musimy także zadbać o odpowiednią liczbę nukleotydów pomiędzy promotorem, pozostałością po miejscu restrykcyjnym i insertem, aby nie doszło do przesunięcia ramki odczytu. Niestety praca z enzymami restrykcyjnymi pozostawia tzw. szwowe nukleotydy (scar nucleotides), których nie możemy usunąć, a które nie mają sensu biologicznego.
Wszystko to sprawia to, że niekiedy bardzo trudno jest dobrać odpowiednie enzymy restrykcyjne do naszych warunków.
Dalszy krok obejmuje sklejanie elementów z użyciem ligazy. Często zdarza się, że po wysianiu transformantów, bez końca testujemy dziesiątki kolonii w poszukiwaniu pozytywnego klonu. Dzieję się tak ponieważ większość plazmidów zostało zamkniętych enzymatycznie bez insertu.
Przygotowanie insertu i plazmidu poddanych obróbce enzymami restrykcyjnymi, duże prawdopodobieństwo powstania fałszywie pozytywnych kolonii, które następnie należy poddać testom sprawia, ze metoda ta trwa bardzo długo od 2 dni do kilku tygodni (Fig2).
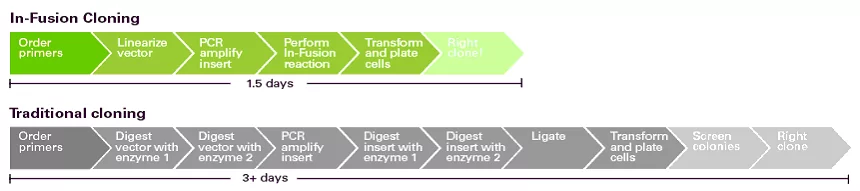
Fig.2 Porównanie długości i etapów metody klonowania opartego na enzymach restrykcyjnych i In-Fusion® [źródło: takarabio .com]
Nomenklatura
Zazwyczaj w procesie klonowania sekwencja nowo wprowadzana nazywa się insertem (insert) lub wstawką a element genetyczny pozwalający na replikację, ekspresję i często z elementem reporterowym to plazmid, wektor (plasmid). Te ostatnie elementy to zazwyczaj koliste cząsteczki DNA, zdolne do autoreplikacji, występujące w bakteriach.
Wspomniane miejsca na plazmidzie to
- ori specyficzne dla organizmu do którego wklonywywany jest plazmid (miejsce początku replikacji)
- promotor jeśli plazmid jest ekspesyjny (zapoczątkowanie transkrycji),
- MCS (multi cloning site) fragment zazwyczaj za promotorem, koncentracji miejsc cięcia dla enzymów restrykcyjnych (pozwalający na wstawienie insertu)
- kaseta oporności na dany antybiotyk (np. Ampicylinę) lub kaseta dla β-galaktozydazy (jako reporter)
W przypadku reportera oporności na antybiotyk pisaliśmy trochę więcej we wpisie – Antybiotyki – cenne narzędzie biotechnologiczne wykorzystywane nie tylko w walce z chorobami w sekcji – jak stosowane są antybiotyki w biotechnologii?
Natomiast kaseta dla β-galaktozydazy pozwala na tzw. blue-white screening kolonii. Enzym jest elementem operonu laktozowego (gen lacZ) i w naturalnych warunkach umożliwia metabolizm laktozy. Natomiast ma także zdolność do rozkładania bezbarwnego związku X-gal (analogu laktozy). Rozkład ten powoduje powstawanie niebieskiego zabarwienia (w procesie powstaje 5-bromo-4-chloro-indoksyl, który następnie spontanicznie dimeryzuje i utlenia się, tworząc jasnoniebieski nierozpuszczalny pigment 5,5′-dibromo-4,4′-dichloro-indigo).
Także komórki poprawnie transformowane plazmidem z nieprzerwaną kasetą dla β-galaktozydazy będą barwiły się na niebiesko. A komórki które nie uległy transformacji będą po dodaniu X-gal białe (tj. naturalnego koloru) (Fig.3).

Fig.3 Przykładowe zdjęcie płytki po analizie kolonii – blue-white screening. [źródło: Wikipedia]
W tym miejscu warto wspomnieć o procesie zwanym α-komplementacją.
Czym jest α-komplementacja?
Gen lacZ (dla β-galaktozydazy) można podzielić na dwie funkcjonalne części: peptyd α (część N-końcowa) i fragment ω (część C-końcowa). Gdy obie części są obecne w tej samej komórce, mogą się wzajemnie uzupełniać, tworząc funkcjonalny enzym β-galaktozydazy, nawet jeśli są wytwarzane z oddzielnych łańcuchów polipeptydowych. Proces ten znany jest jako α-komplementacja (α-Complementation).
W praktyce, α-komplementacja jest szeroko stosowana we wspomnianych wyżej analizach przesiewowych wyrośniętych kolonii tj. blue-white screeningu.
Ponieważ procedury klonowania wykorzystują ligazy, często zdarza się, że pomimo iż klony wyrastają na podłożu selekcyjnym. nie zawierają wstawki (są negatywne w analizie kolonijnego PCR, colony PCR). Są tzw. puste. Dzieje się to dlatego, że enzym liguje końce plazmidu przed tym jak dojdzie do komplementarnego złączenia insert-plazmid.

Zapytaj nas o SapphireAmp, premiks zoptymalizowany do przeprowadzania kolonijnego PCR.
Metoda wykorzystująca α-komplementację pozwala na rozróżnienie rekombinowanych i nierekombinowanych plazmidów w eksperymentach klonowania. Jak się to dzieje?
Konieczne są dwa warunki:
- szczep transformowany E. coli, musi mieć mutację w swoim własnym genie lacZ (lacZΔM15), co czyni go niezdolnym do samodzielnego wytwarzania funkcjonalnej β-galaktozydazy (może jednak wytwarzać fragment ω β-galaktozydazy). Warunki takie spełniają komórki JM109, DH5α i XL1-Blue. (Warto wspomnieć, że złoża posiewowe nie powinny zawierać w składzie glukozy)
- plazmid musi być odpowiednio przysposobiony do metody. Musi zawierać gen lacZ, jednak jego region α musi być przecięty sekwencją MCS – czyli miejscem wstawiania insertu, czyli np. pUC19 i pBluescript.
Podczas klonowania insert zostanie wstawiony w MCS w środek regionu α, genu lacZ plazmidu. Powoduje to błędy w komplementacji β-galaktozydazy. W rezultacie komórki zawierające rekombinowane plazmidy nie wybarwiają się na niebiesko ponieważ, nie wytwarzają funkcjonalnego enzymu (Fig.4). Komórki zaś z pustym plazmidem są niebieskie. Czyli odwrotnie do sytuacji gdy kaseta dla β-galaktozydazy służy jako reporter (np. drożdże).
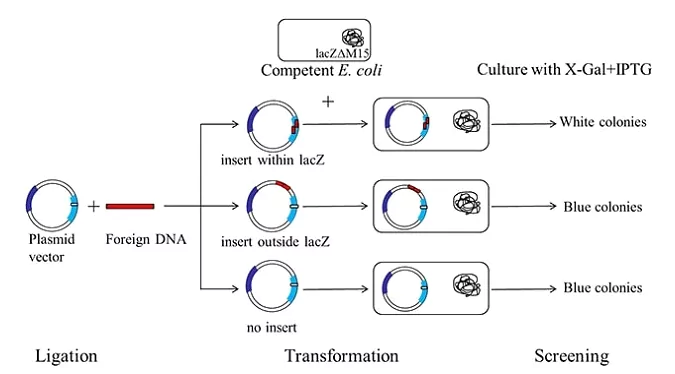
Fig.4. Schemat wykorzystania zjawiska α-komplementacji w analizie efektywności klonowania (blue-white screening) [źródło: sigmaaldrich. com]
Czym jest klonowanie tzw. „seamless”? Przykłady systemów.
Pomimo swojego ogromnego znaczenia, klonowanie molekularne nie jest pozbawione wyzwań, o kilu wspomnieliśmy omawiając klasyczną technikę klonowania.
Proces ten może być wymagający technicznie i czasochłonny, wymagając starannej optymalizacji warunków eksperymentalnych. Co więcej, wybór wektora i organizmu gospodarza może mieć wpływ na wydajność i powodzenie eksperymentów klonowania, dlatego przez długi czas starano się opracować technikę klonowania bardziej przyjazną dla użytkowników i efektywniejszą.
Tak powstały systemy do klonowania „seamless”, czyli takie nie pozostawiające nukleotydów szwowych (pozostałości po działaniu restryktazami).
Ponad to ten rodzaj klonowania umożliwia badaczom uniezależnienie od sekwencji restrykcyjnych a w konsekwencji swobodny dobór wektorów i wstawek, a nawet tworzenie fuzyjnych sekwencji (fuzyjnych białek rekombinantów). Poniżej zostaną omówione 3 metody seamless – Gibson Assembly, klonowanie z użyciem enzymów restrykcyjnych (RE) typu IIS i In-Fusion® (Fig.5). Każda z nich wykorzystuje PCR do amplifikacji insertów, ale mechanizmy łączenia insertów z wektorami są odmienne.
Fig.5. Zestawienie metod nowej generacji typu seamless
Gibson Assembly
Metoda Gibsona (Gibson et al. 2009) wykorzystuje mieszaninę trzech enzymów (egzonukleazy, ligazy i polimerazy DNA) do przejścia od liniowych fragmentów DNA do gotowego plazmidu. Podobnie jak w przypadku innych metod klonowania bezszwowego (seamless), wstawki są amplifikowane metodą PCR, tak aby zawierały homologiczne flanki ze zlinearyzowanym plazmidem lub innymi sąsiednimi fragmentami. Zaleca się, aby długość tych elementów zawierała się pomiędzy 15-25 nt i aby ją dobrać eksperymentalnie. Po złożeniu reakcji , 5′ egzonukleaza hydrolizuje DNA w celu utworzenia lepkich końców. Po odnalezieniu przez insert i plazmid komplementarnych końców, polimeraza DNA (o ograniczonej aktywności proofreading) uzupełnia luki pomiędzy elementami konstruktu a ligaza scala wszystko w jedność (in vitro) (Fig.5.).
Nowsza wersja metody NEBuilder HiFi DNA Assembly wykorzystuje tą samą technologię, ale znajdująca się w premiksie polimeraza jest wysokiej wierności. Przez co powstaje mniej mismatchy w odcinkach przed i za insertem. W przypadku obu wersji metody Gibsona, do ostatecznego scalenia wykorzystywana jest aktywność ligazy będącej w mieszaninie od początku. Obecność ta sprawia, że dochodzi do zamykania pustych plazmidów i powstawania kolonii fałszywie pozytywnych, które musimy odrzucić podczas kolonijnego PCR.
Sprawia to, że metoda jest mniej efektywna przy wysokoprzepustowym klonowaniu i tworzeniu fuzyjnych konstruktów.
Klonowanie RE typu IIS
Enzymy restrykcyjne typu IIS (RE) tną DNA poza swoimi miejscami restrykcyjnymi, co sprawia, że nie tworzą się tzw. szwowe nukleotydy jak w przypadku klasycznego klonowania (Toth et al. 2014). Niemniej etapy metody są bardzo podobne do klonowania z użyciem zwykłych enzymów restrykcyjnych. Wstawki i wektory są zaprojektowane w taki sposób, że miejsce restrykcyjne jest usuwane przez sam enzym (Fig.5.).
Flanki mogą być specjalnie zaprojektowane, aby zapewnić kierunkowe (sensowne) wstawienie jednego insertu lub wielu fragmentów w tej samej reakcji. Takie wykorzystanie metody jest jednak mało wydajne i może wymagać dodatkowych manipulacji i wstawiania lub usuwania miejsc restrykcyjnych w obrębie plazmidu i insertów.
Ten rodzaj klonowania wykorzystuje dostępny na rynku Golden Gate Assembly. Zawiera mieszaninę enzymów restrykcyjnych typu IIS RE oraz ligazę DNA T4.
Czym jest klonowanie In-Fusion?
Technologia klonowania In-Fusion® (Irwin et al. 2012) w odróżnieniu od omówionych metod seamless zapewnia klonowanie produktów PCR bez ligacji in vitro. System umożliwia klonowanie dowolnego insertu w każdy zlinearyzowany plazmid, niezależnie od locus. Ponieważ metoda ta nie wykorzystuje restrykaz, nie ma potrzeby enzymatycznej obróbki fragmentu PCR (np. trawienie restrykcyjne, fosforylacja), a ponieważ w mieszaninie nie ma ligazy, obecność kolonii „tła” (fałszywie pozytywnych) jest niezwykle niskie (tj. powstawanie pustych wektorów). Typowe problemy wynikające z nieefektywności ligacji są również wyeliminowane, umożliwiając pominięcie etapów subklonowania, nawet w przypadku dużych wektorów i wstawek (plazmidy adenowirusowe, inserty do 34 kb).
Wektory liniowe mogą być przygotowywane za pomocą trawienia restrykcyjnego lub w wyniku PCRu, oferując możliwości prawdziwego klonowania niezależnego od sekwencji. Reakcja in-Fusion® jest jednoetapowa i trwa jedynie 15 minut.
Podobnie jak metoda Gibsona opiera się na homologii fragmentów między plazmidem a wstawką. W przeciwieństwie jednak do niej, wspomniane odcinki zawsze mają długość 15 nt, nie trzeba zatem optymalizować warunków reakcji a każda reakcja jest kierunkowa i dokładna. W mieszaninie działa egzonukleaza 3’, hydrolizująca fragmenty DNA i tworząca przez to kompatybilne ze sobą końce na zasadzie komplementarności. W takiej formie materiał jest transformowany do komórek kompetentnych, które ligują konstrukt (in vivo) chroniąc przed wprowadzeniem jakichkolwiek błędów sekwencji lub niedopasowaniem nukleotydów.
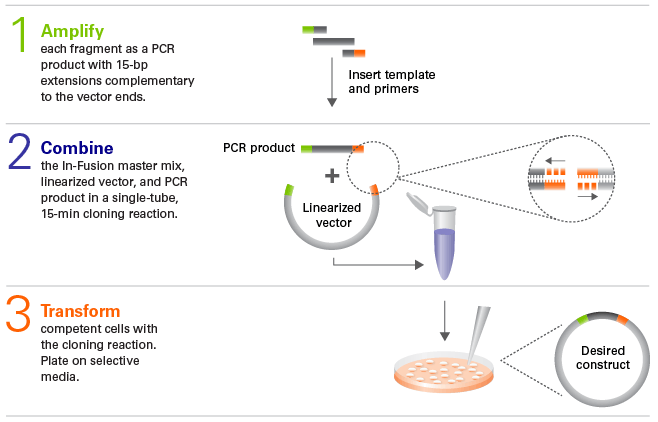
Fig.6. Schemat reakcji In-Fusion [źródło: takarabio .com]
Takie rozwiązanie zapewnia wysoką dokładność i efektywność na poziomie 95% usprawniając pracę. Metoda sprawdza się także w klonowaniu wysokoprzepustowym i tworzeniu fuzyjnych konstruktów (multiklonowanie, multicloning).
Klonowanie In-Fusion jest niezwykle wszechstronne. Dzięki łatwemu do przestrzegania protokołowi, który zapewnia wysoką wydajność, można z powodzeniem wykonać szeroką gamę zastosowań, a wszystko to przy użyciu tego samego zestawu (Fig.7).
Pozytywne klony w 2 dni z 95% wydajnością