CRISPR/Cas9 – historia, zastosowania i nowe metody syntezy ssDNA

Emmanuelle Charpentier i Jennifer A. Doudna zostały w 2020 roku uhonorowane Nagrodą Nobla w dziedzinie chemii za rozwój metody CRISPR/Cas9, nazywanej potocznie precyzyjnymi nożyczkami genetycznymi. Należy jednak pamiętać, że zainteresowanie wielu badaczy oraz jednostek naukowych na całym Świecie tym niezwykle precyzyjny narzędziem do edycji genów sięga późnych lat 80 tych XX wieku.
Pierwsze wzmianki literaturowe dotyczące tych zgrupowanych, regularnie rozproszonych, krótkich, powtarzających się sekwencjach palindromowych, bo tak należy tłumaczyć z angielskiego rozwinięcie skrótu CRISPR (ang. Clustered Regularly-Interspaced Short Palindromic Repeats) datuje się na rok 1987. Wtedy to też japoński uczony Yoshizumi Ishino odkrył, że bakterie Escherichia coli posiadają w swoim materiale genetycznym, w ściśle określonym miejscu, kilka powtarzających się sekwencji . Nie znano wtedy jeszcze funkcji jakie pełnią one w genomie bakteryjnym. Podobne zależności zaobserwował hiszpański mikrobiolog Francisco Mojica. Badając materiał genetyczny słonowodnych archeonów z gatunku Haloferax mediterranei zidentyfikował on występowanie 14 regularnie powtarzających się, trzydziestonukleotydowych, palindromowych sekwencji DNA. Te powtarzające się sekwencje rozdzielone były pozornie przypadkowymi, trzydziestopięcionukleotydowymi sekwencjami DNA, które nazwano „spacer’ami”.
Dopiero w 2005 roku, trzy niezależne zespoły badawcze odkryły, że te pozornie przypadkowe sekwencje DNA pełnią bardzo istotne funkcje w odpowiedzi organizmów bakteryjnych na inwazję bakteriofagów. W dużym uproszczeniu, są to niejako „zdeponowane” w genomie bakteryjnym fragmenty wybranych genów wirusów bakteryjnych, które po transkrypcji na mRNA, odpowiedniej obróbce potranskrypcyjnej i przyłączeniu tzw. tractRNA są przekształcane w sgRNA. Te krótkie sekwencje RNA nazywane sygnaturą (wzorcem) wirusa wraz z białkiem Cas (ang. CRISPR associated protein) są następnie wykorzystywane w mechanizmach obronnych komórek bakteryjnych .
Intensywny postęp badań nad metodą CRISPR/Cas9 spowodował, że obecnie jest to dość powszechne i niesamowicie precyzyjne narzędzie pozwalające na niemal nieograniczoną edycję genomów. W 2018 roku w mediach pojawiły się doniesienia o narodzinach pierwszych bliźniąt z genami zmodyfikowanymi dzięki zastosowaniu CRISPR/Cas9. Jak ogłosił wówczas autor eksperymentu prof. He Jiankui, biofizyk z Południowego Uniwersytetu Nauki i Technologii w Shenzhen (Chiny), modyfikacja miała polegać na usunięciu genu CCR5 u embrionów uzyskanych dzięki zapłodnieniu in vitro, tak by uodpornić je na zakażenie wirusem HIV. Brak jest jednak oficjalnych dowodów naukowych potwierdzających przeprowadzenie tego eksperymentu.
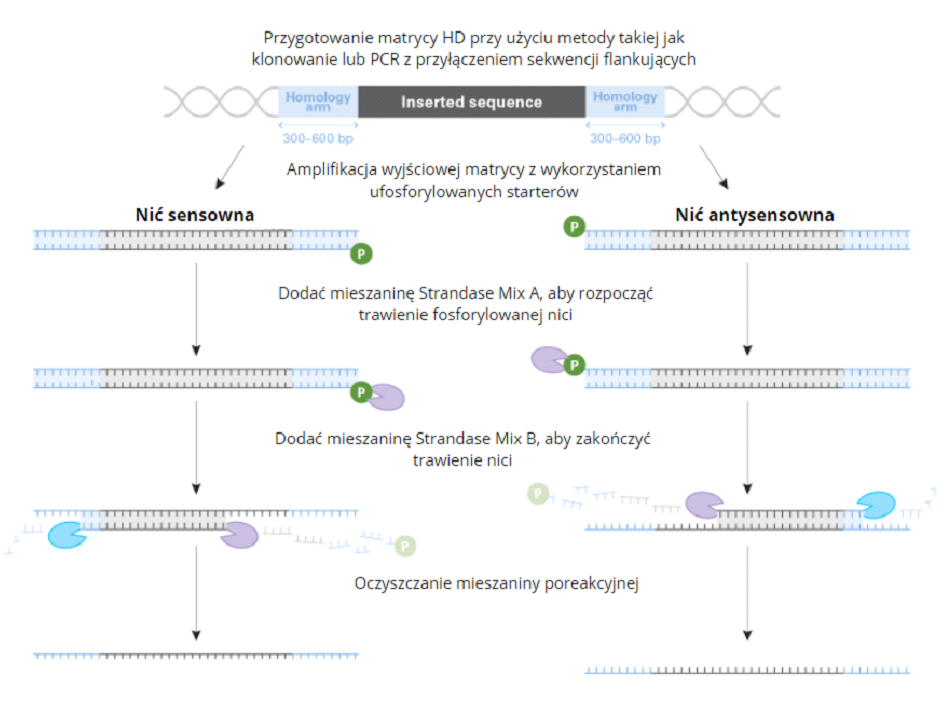
Jak wspomniano, CRISPR/Cas jest potężnym narzędziem do edycji genomów. Ponadto, jest to metoda stosunkowo tania, wydajna i relatywnie prosta w wykonaniu. W eksperymentach mających na celu wyciszenie poszczególnych genów tzw. knock-out genów, do przeprowadzenia eksperymentu (w dużym uproszczeniu) konieczna jest znajomość docelowej sekwencji, odpowiednio zsyntetyzowany sgRNA oraz sprzężone z nim białko z rodziny Cas. Generowane w wyniku działania endonukleaz z rodziny Cas „rozcięcia” obu nici DNA są następnie naprawiane 1 wykorzystaniem mechanizmu łączenia niehomologicznych zakończeń (NHEJ, ang. non homologous end joining). Mechanizmy te generują liczne błędy w kodzie genetycznym, w wyniku których dochodzi do zmiany ramki odczytu lub mutacji, które wpływają na funkcjonalność genów lub eksprymowanych białek. Odmienne mechanizmy naprawy DNA wykorzystywane są w eksperymentach polegających na wprowadzaniu do sekwencji docelowych określonych polimorfizmów pojedynczych nukleotydów (SNP, ang. single nucleotide polymorphism), krótkich lub długich insercji. Knock-in genów, bo tak nazywane są te eksperymenty, poza sgRNA oraz dostępnością białka Cas wymaga obecności odpowiednio zsyntetyzowanych matryc naprawczych HDR (ang. homology directed repair template). Matryce te poza sekwencjami, które chcemy wprowadzić i/lub zmienić powinny zawierać odcinki flankujące o długości ok. 300-600 pz, które są homologiczne z sekwencjami docelowymi w genomie modyfikowanego gospodarza. Dostępność tak przygotowanych matryc HDR pozwala na uruchomienie mechanizmów naprawy DNA wykorzystujących mechanizmy rekombinacji homologicznej (HDR, ang. homology directed repair) .
Matryce HDR mogą być dostarczane do modyfikowanych komórek w postaci dsDNA (double-stranded DNA – plazmidy lub formy zlinearyzowane) lub w formie ssDNA (single stranded – jako AAV lub sekwencje liniowe). Badania przeprowadzone przez Theodore L. Roth i wsp. wykazały, że dla relatywnie długich insercji najbardziej efektywną postacią matryc HDR jest ich forma ssDNA. Takie jednoniciowe sekwencje charakteryzują się zdecydowanie mniejszą toksycznością dla komórek gospodarza, co znajduje odzwierciedlenie w wielkości populacji żywych komórek po dostarczeniu matryc. Ponadto, zastosowanie ssDNA znacząco zmniejsza prawdopodobieństwo wystąpienia losowych lub niezgodnych z celem integracji w genomie modyfikowanych organizmów. Stosowane powszechnie metody syntezy ssDNA mają jednak pewne ograniczenia. Dla jednoniciowych DNA otrzymywanych z wykorzystaniem AAV największym ograniczeniem okazuje się być pojemność AAV. Limit wielkości dla matryc pozyskiwanych tym sposobem wynosi ok. 2,5 kpz. Ponadto, jest to metoda czasochłonna i wymaga relatywnie dużych nakładów pracy. Dłuższe sekwencje ssDNA pozyskać można z wykorzystaniem metody chemicznej syntezy kwasów nukleinowych. Niestety dla sekwencji ssDNA>200pz cena usługi wzrasta drastycznie wraz z długością zamawianej matrycy. Ponadto, czas oczekiwania na zamawiane sekwencje wynosi nawet 20 dni roboczych.
Wychodząc naprzeciw oczekiwaniom i potrzebom swoich klientów firma TakaraBio opracowała innowacyjny zestaw pozwalający na szybką, wydajną i tanią syntezę ssDNA, których wielkość może wynosić nawet 5 000 pz. Zestaw Guide-it™ Long ssDNA Production System v2 jest prosty w zastosowaniu, a czas niezbędny na otrzymanie odpowiedniej ilości ssDNA można ograniczyć do kilku godzin. Do przeprowadzenia syntezy dowolnej matrycy HDR wymagana jest jedynie wzorcowa, dwuniciowa matryca HDR, którą pozyskać można poprzez klonowanie In-Fusion lub reakcję PCR z przyłączeniem homologicznych sekwencji flankujących do docelowej sekwencji będącej obiektem zainteresowania. Tak przygotowaną dwuniciową matrycę należy jedynie zamplifikować z wykorzystaniem ufosforylowanych sekwencji starterowych (rozłączna reakcja dla starterów 5’P – i 3”P-), a następnie poddać działaniu koktajli enzymatycznych degradujących jedną z nici. Oczyszczony produkt można wykorzystać do elektroporacji komórek gospodarza wraz z sgRNA oraz białkiem Cas. Uproszczony schemat działania zestawu Guide-it™ Long ssDNA Production System v2 przedstawiono na Figurze 1.